UNETR Transformers for 3D Medical Image Segmentation
3D Input이미지를 ViT + 3D U-NET 인 Method로 segmentation을 진행한 모델 ⇒ UNETR
기존 3D Segmentation 방식
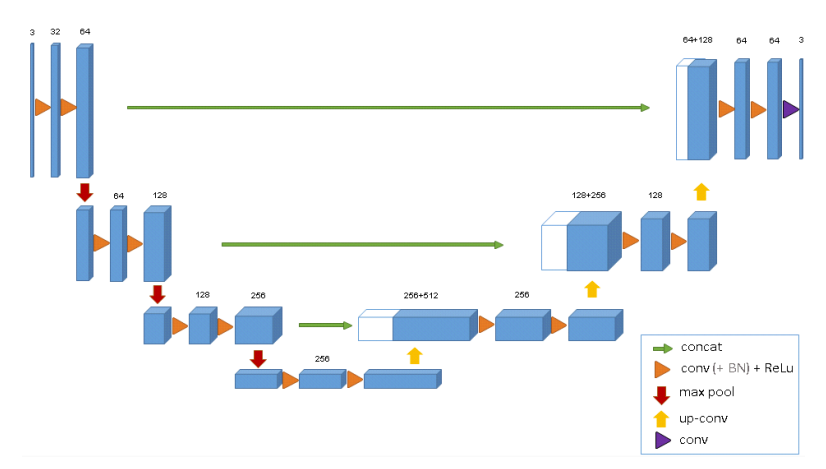
3D U-Net
UNETR 이전의 3D 이미지를 Segmentation하는 방법은 위의 이미지와 같다.
위 이미지는 3D U-Net 모델의 구조인데 이는 기존의 2D 이미지를 Segmentation하는 모델인 U-Net을 기반으로 하고 있으며 다른 점은 input 데이터의 shape이 3차원(Voxel)이라는 점이다.
하지만 이러한 방법의 단점으로는 장거리 공간적 의존성을 학습하기 어렵다는 단점이 있다.
UNETR
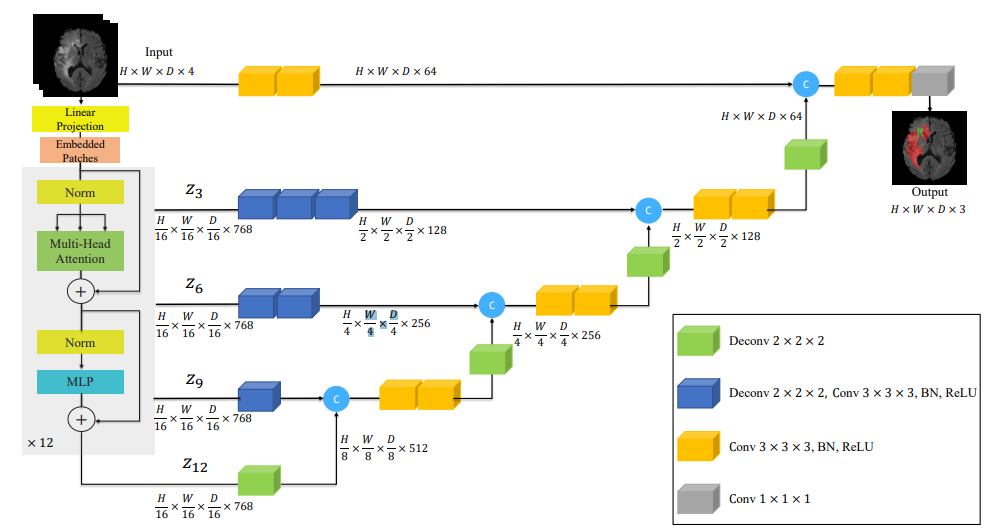
UNETR
앞서 말했듯 기존 3D segmentation에서의 단점을 해결하기 위해 Transformer를 사용하기로 합니다.
모델 구조에서 인코더 부분에 ViT를 적용하는 방식을 사용합니다.
따라서 인코더에서는 ViT를 이용해 전역 정보를 파악하고 U-형 네트워크 구조와 스킵 연결을 이용해 디코더와 결합하는 구조를 가지게 됩니다.
이러한 구조를 통해 기존의 3D segmentation의 문제인 장거리 의존성 학습 문제를 해결하게 되었습니다.
| 단계 | Shape | 설명 |
|---|---|---|
| 입력 | MRI 다채널 3D 입력 | |
| 패치 분할 | 512개의 |
|
| 패치 임베딩 | 각 패치를 768차원으로 투영 | |
| 위치 임베딩 추가 | 공간 정보 보존 | |
| 트랜스포머 출력 | 12 계층을 거친 후 동일 크기 유지 | |
| 3D 복원 | 3D 텐서로 복원 |
|
| 디코더 및 최종 출력 | 클래스별 세그멘테이션 결과 |
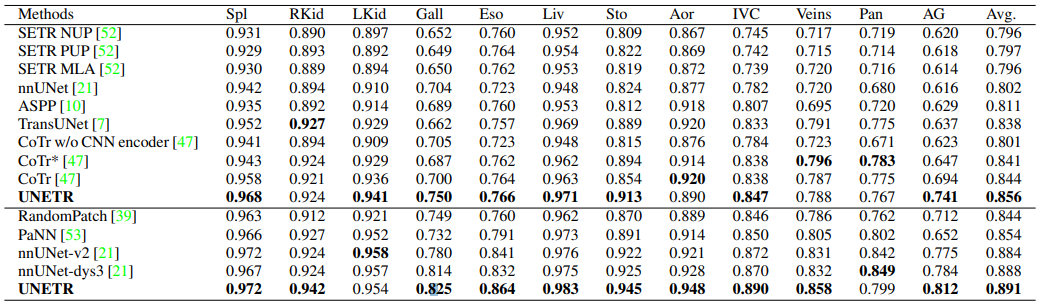
- BTCV (복부 CT 스캔 데이터셋)
- MSD (뇌종양 Segmentation 데이터셋)
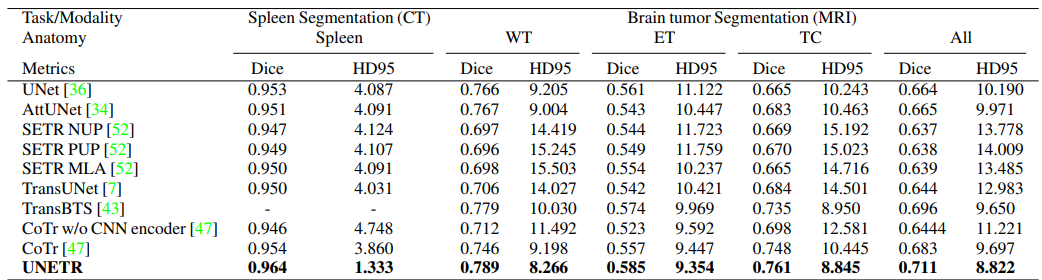
두 가지 데이터셋을 이용해 학습 후 평가한 결과 기존에 존재하던 모델에 비해 더 좋은 성능을 보여준 다는 것을 확인할 수 있었고 결과적으로 UNETR은 기존 모델과 비교해 다음과 같은 강점이 있다는 것을 알았습니다.
- Transformer를 이용한 전역 및 지역 의존성 모두 학습 가능
- 장거리 의존성이 중요한 경우에 더 높은 성능의 정확도
- 작은 장기 분할에서 기존 모델 대비 월등한 성능
-
추가연구
-
기존의 UNETR의 디코더를 말고 다른 구조의 디코더를 사용하면 성능 변화가 어떨까?
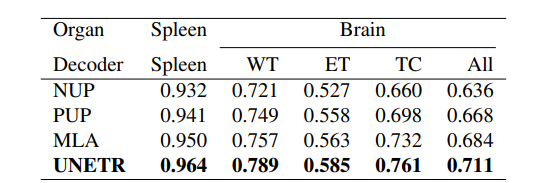
MSD 데이터 셋을 이용해 여러 디코더를 평가한 결과 다른 디코더 보다 기존에 적용한 UNETR의 디코더가 더 성능이 좋은 것을 알 수 있었다.
-
ViT input에 사용되는 패치 사이즈를 조절하면 어떻게 될까??
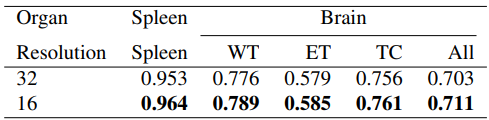
패치 사이즈를 32 → 16으로 낮추었더니 성능이 높아진 것을 확인 가능하다.
하지만 패치 사이즈를 낮추면 시퀀스가 길어져 메모리를 많이 사용한다는 단점이 있다. -
다른 모델들과 계산 복잡도 비교
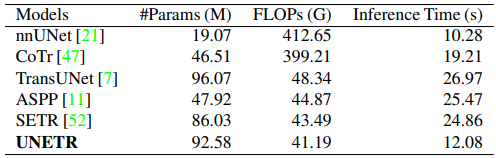
추론 시간은 nnUNet 다음으로 짧으며, 다른 트랜스포머 기반 모델보다 훨씬 빠른 것을 알 수 있었다.
-
-
UNETR++
U-NETR 업그레이드 버전
-
Swin_UNETR
ViT가 세밀한 영역을 잡지 못하는 것을 보완한 모델